pymatgen 使用¶
约 3129 个字 1111 行代码 1 张图片 预计阅读时间 24 分钟
介绍¶
-
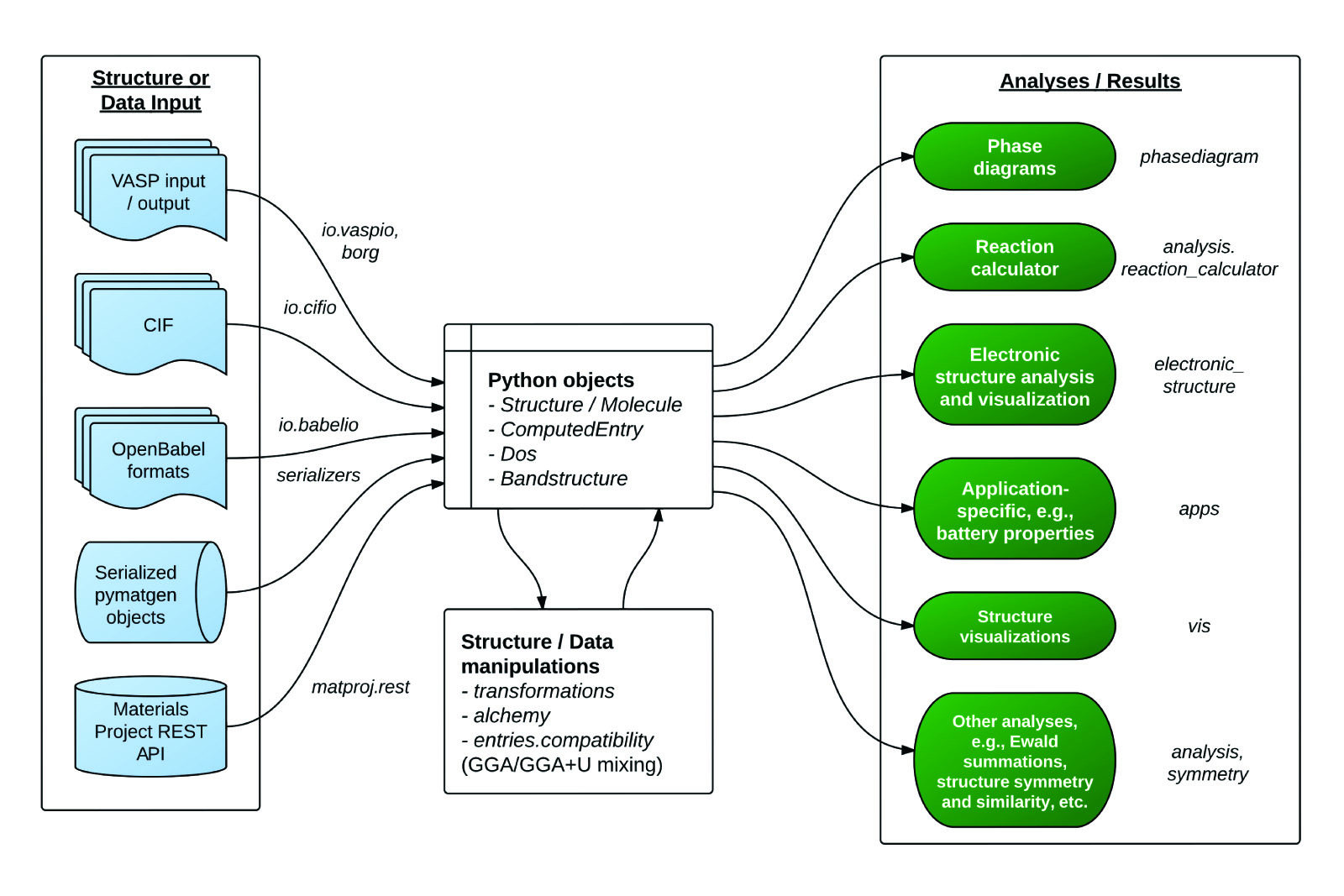
用于表示 Element、Site、Structure、Molecule 的高度灵活的类
-
文件输入/输出支持广泛,如 VASP、ABINIT、CIF、Gaussian、XYZ 等(主要依靠 Open Babel 包)
-
强大的分析工具,包括生成相图、Pourbaix 图、扩散分析、反应等
-
电子结构分析,如态密度和能带结构
-
集成 Materials Project REST API、Crystallography Open Database 等其他外部数据源
-
代码文档详细
参考资料¶
-
pymatgen notebook:GitHub - yw-fang/pymatgen-notebook
-
pymatgen 实例代码:GitHub - materialsvirtuallab/matgenb
-
Materials Project 官方文档:Materials Project Documentation
- Materials Project 推荐赝势:Pseudo-potentials - Materials Project Documentation
- 含 MP 在计算中所采用的具体参数及其说明(如截断能为 520eV 是由元素周期表所有元素中最大截断能的 1.3 倍得到的): Materials Methodology - Materials Project Documentation
-
GitHub - computron/pymatgen_tutorials: Tutorials for using the pymatgen library
-
Material Project Workshop:
- 2021:The Materials Project Workshop
- 2018~2020:Releases · materialsproject/workshop
- 2017:GitHub - materialsproject/workshop-2017: Assets for the 2017 Materials Project workshop
- 2016:GitHub - materialsproject/workshop-2016: Assets for the Materials Project workshop in Aug 2016
- 注:Workshop 2020 和 2021 的内容绝大部分相似,lesson3 分别为表面和界面;Workshop 2018 和 2019 的内容相似(对 atomate 的讲解稍微详细些)
安装¶
-
安装
# 稳定版本
pip install -U pymatgen
# 开发版本
pip install -U git+https://github.com/materialsproject/pymatgen
-
赝势目录设置
-
change log(代码 bug 修复,新功能添加等,可以关注)
-
兼容性:需对进行
from pymatgen import xxx修改(v2022.0.0 版本开始)
# 旧
from pymatgen import IMolecule, IStructure, Molecule, Structure
from pymatgen import PeriodicSite, Site
from pymatgen import Composition
from pymatgen import Lattice
from pymatgen import DummySpecie, DummySpecies, Element, Specie, Species
from pymatgen import SymmOp
from pymatgen import ArrayWithUnit, FloatWithUnit, Unit
from pymatgen import Orbital, Spin
from pymatgen import MPRester
# 新
from pymatgen.core.structure ...
from pymatgen.core.sites ...
from pymatgen.core.composition import Composition
from pymatgen.core.lattice import Lattice
from pymatgen.core.periodic_table ...
from pymatgen.core.operations import SymmOp
from pymatgen.core.units ...
from pymatgen.electronic_structure.core ...
from pymatgen.ext.matproj ...
# 或如下写法;因为 pymatgen/core/__init__.py 中已全部写入上述模块导入内容
from pymatgen.core import Structure, Composition ...
使用¶
-
pymatgen 的包 (package、subpackage) 是目录, 模块 (module、submodule) 是文件; import 既可以导入包和子包, 也可以导入模块和子模块; ug-materials-simulation/pymatgen/包和模块结构.ipynb at master · xiangzhouzhang/ug-materials-simulation · GitHub
-
查看 pymatgen 相关信息
import pymatgen.core
import sys
sys.version # 查看 Python 版本
pymatgen.core.__version__ # 查看 pymatgen 版本
pymatgen.core.__file__ # 查看 pymatgen 安装路径
# 获取 .pmgrc 配置文件内容
from pymatgen.core import SETTINGS
PMG_VASP_PSP_DIR = SETTINGS.get("PMG_VASP_PSP_DIR")
# 可解析压缩文件路径
from monty.os.path import zpath
path = ...
path = zpath(path)
- pymatgen 支持的构型文件格式
FileFormats = Literal[
"cif",
"poscar",
"cssr",
"json",
"yaml",
"yml",
"xsf",
"mcsqs",
"res",
"pwmat",
"aims",
"",
]
- pymatgen 中可指定的泛函类型
FUNCTIONAL_CHOICES= ['PBE', 'PBE_52', 'PBE_54', 'LDA', 'LDA_52', 'LDA_54', 'PW91', 'LDA_US', 'PW91_US', 'Perdew-Zunger81']
-
MP 晶体 DFT code 用的是 VASP,分子用的是 Q-Chem
-
MSONable类:MSON(Monty JSON);MSONable 对象必须实现as_dict()方法,该方法须返回可序列化为 JSON 的字典,且须支持无参数。静态方法from_dict(),从as_dict()方法生成的字典中重建对象。as_dict()方法应该包含@module和@class键,这将允许 MontyEncoder 动态反序列化该类。 -
monty 包:对 json/yaml/msgpack 等文件格式进行 serialization
工作流¶
pymatgen 典型工作流
CLI¶
- 不是太好用,建议直接写脚本
pmg subcommand -h # 查看子命令帮助
# 分析当前路径,会将生成的数据打包压缩成文件
pmg analyze .
# -f 单个文件;--filenames 可多个文件
# 查看结构空间群信息
pmg structure -s 0.00001 -f POSCAR
# 构型文件转换,功能有限
# Supported formats include POSCAR/CONTCAR, CIF, CSSR, etc.
pmg structure --convert --filenames POSCAR *.cif
# 可视化构型;需安装 vtk 包;会报错,不建议
pmg view POSCAR
- pymatgen 的 cli 可以使用 argcomplete 库(
pyamtgen/cli/pmg.py;用于 cli 命令的补全)
try:
import argcomplete
argcomplete.autocomplete(parser)
except ImportError:
# argcomplete not present.
pass
操作 structure¶
- 包括创建、保存、分析与变化操作
from pymatgen.core.composition import Composition
from pymatgen.core.lattice import Lattice
from pymatgen.core.structure import Structure
from pymatgen.symmetry.analyzer import SpacegroupAnalyzer
# 标准方法
structure = Structure(
Lattice.cubic(4.2),
["Cs", "Cl"],
[[0, 0, 0], [0.5, 0.5, 0.5]],
)
# 利用空间群对称性创建结构
structure = Structure.from_spacegroup(
"Fm-3m",
Lattice.cubic(3),
["Li", "O"],
[[0.25, 0.25, 0.25], [0, 0, 0]],
)
# 保存成其他文件格式
structure.to(filename="POSCAR", fmt="poscar")
# 不提供 filename 参数,返回 string
structure.to(fmt="poscar")
# 只提供 filename 参数,会自动识别其格式
structure.to(filename="POSCAR")
structure.to(filename="CsCl.cif")
# 从 str 或文件中读取结构
structure = Structure.from_str(open("CsCl.cif").read(), fmt="cif")
structure = Structure.from_file("CsCl.cif")
# 改变位点元素种类
structure[1] = "F"
# 改变位点元素种类和坐标
structure[1] = "Cl", [0.51, 0.51, 0.51]
# 元素替换
structure["Cs"] = "K"
# 生成无序结构
# 与 SQS 是不同的概念
# 部分占据的无序结构无法保存成 POSCAR
structure["K"] = "K0.5Na0.5"
# structure 类似 list,支持大部分的 list 方法
# reverse, append, extend, pop, index, count
structure.reverse()
structure.append("F", [0.9, 0.9, 0.9])
# 超胞构建
structure * 2
structure * (2, 2, 2)
structure.make_supercell([2, 2, 2])
pymatgen 的许多类都有 as_dict() 方法和 from_dict() 静态方法的实现。虽然 python 确实提供了 pickling 功能(实现对象序列化和反序列化的方式),但 pickle 在代码修改方面往往是非常脆弱的。as_dict() 提供了一种以更稳健的方式保存工作的方法,且更容易阅读。将 object 输入某些数据库,如 MongoDB,也特别有用。as_dict() 规范是由 monty 库(pymatgen 产生的一个通用 python 补充库)提供的。
可使用 PyYAML 包的 yaml 代替上述任何 json 命令来创建 yaml 文件(JSON 格式效率高,读写速度快,但可读性差;YAML 解析速度慢,但更适合人类阅读)
更精细地控制从文件读取解析结构,可以使用特定的 io 包。这些包也提供了导出文件格式的方法。
from pymatgen.io.cif import CifParser
parser = CifParser("mycif.cif")
structure = parser.get_structures()[0]
from pymatgen.io.vasp.inputs import Poscar
poscar = Poscar.from_file("POSCAR")
structure = poscar.structure
from pymatgen.io.xyz import XYZ
from pymatgen.io.gaussian import GaussianInput
xyz = XYZ.from_file('methane.xyz')
gau = GaussianInput(xyz.molecule,
route_parameters={'SP': "", "SCF": "Tight"})
gau.write_file('methane.inp')
Entry¶
- Entry 的最基本形式:一个构型成分 + 对应计算的能量(可包含其他输入或计算数据);可作为 pymatgen 创建相图的计算数据集
计算输入输出管理¶
-
pymatgen.io 模块包含一些计算软件(主要是 VASP)的输入文件编写、解析与输出文件解析子模块
-
输入管理的核心类是
InputSet。InputSetobject 包含计算输入文件所需的所有数据。具体来说,write_input()方法,可将所有文件写到指定位置。InputGenerator类可以看作是完成特定计算任务的 recipe,而InputSet则包含这些 recipes 以应用于特定体系或结构 -
也可以使用
InputSet.from_directory()从计算目录中构建 pymatgenInputSet -
许多解析输出文件的类继承自
InputFile,其提供了一个读写文件的标准接口
其他¶
- 生成元素置换后的非等同结构
- 借助 bsym 包;适用于复杂结构,如金属间化合物;可理解为置换找到的非等同原子中等同原子的第一个原子
- bsym_examples
from bsym.interface.pymatgen import unique_structure_substitutions
subs_structures = unique_structure_substitutions(
structure=structure,
to_substitute="Nb",
site_distribution={"Al": 2, "Nb": 14},
verbose=True,
show_progress=True,
)
- 解析 VASP 计算目录:Automated DFT - The Materials Project Workshop
from atomate.vasp.drones import VaspDrone
drone = VaspDrone()
task_doc = drone.assimilate(path=...)
task_doc.keys()
- 静电势能计算
- 【Pymatgen学习 2】Ewald方法计算静电能
- Ewald Summation - Qijing Zheng
- EwaldSummation 是 pymatgen 库中的一个类,用于计算离子晶体的 Ewald 总能量。Ewald 总能量是一种用于处理带电体系的长程库仑相互作用的技术,通常用于计算固体材料中的电势能。该方法将总能量分解为实空间、倒空间、点电荷修正和偶极修正部分,并进行相应的求和计算
求助:过渡态计算新版pymatgen中找不到iddp插值方法 - 第一性原理 (First Principle) - 计算化学公社
# 过渡态 NEB
from pymatgen.analysis import transition_state
from pymatgen.analysis.diffusion.neb.pathfinder import IDPPSolver
# pymatgen defects 扩展包不好用,不值得深入了解
from pymatgen.analysis.defects.generators import SubstitutionGenerator
subs = SubstitutionGenerator(structure, "Bi")
# 晶界相关
from pymatgen.core.interface import GrainBoundary, GrainBoundaryGenerator
# 待了解
read_neb()
MITNEBSet class
相关问题¶
-
pymatgen 如何获取可用的 POTCAR 种类;较难:一般是指定泛函类型
-
pymatgen structure 如何通过 structure 来生成 potcar?解决方法:通过 Poscar 类得到 structure 的元素种类,之后与 PBE 泛函的元素进行比对,之后用 Potcar 类写入 POTCAR(生成新的之前需删掉原来的 POTCAR 文件)
-
pymatgen 中的 LLL reduction 是什么含义
-
pymatgen 键长计算(并非只是简单的计算原子对之间的距离)
-
pymatgen 保存成 POSCAR 时,对元素类型进行排序(是否可自定义);不方便,建议用 ase ✅ 2024-10-23
-
to()或Poscar类中的write_file()方法无直接排序参数 ✅ 2024-10-23 -
Structure类中的sort()方法中的key参数可以是哪些值(不深挖) ✅ 2024-10-23
-
常用模块¶
pymatgen.core¶
- pymatgen 核心模块,常用的子模块有:
structure、sites、lattice、composition、periodic_table
structure¶
-
晶体结构模块
-
Structure 类继承自 IStructure
-
Structure 与 Molecule 类的区别
- Molecule 类的基本参数为
species、coords,Structure 还需指定lattice参数 - Molecule 类的
coords参数值需是笛卡尔坐标形式,Structure 类可以是笛卡尔和分数两种坐标形式 - Molecule 本质上是 Site objects 的列表;Structure 本质上是 PeriodicSites objects 的列表;可以像 list 一样进行操作
- Molecule 类的基本参数为
-
查看方法是否会直接修改对象本身:检查是否有
in_place参数及注释是否有 "in place" 字样(“原地” 的意思) -
Structure 类无
wrap()方法(根据 PBC 条件将胞外原子移至胞内),ASE 有:pymatgen - What Does the coordinate list next to the cartesian coordinates of an atom represent in neighbor_list - Stack Overflow -
复杂结构无法通过
to_primitive()方法(以及 vaspkit)将单胞转化成原胞 -
Structure 类相关属性和方法:
from pymatgen.core.structure import Structure
# 属性
index # 原子位点序号
num_sites # 原子数;int
composition.num_atoms # 原子数;float
n_elems # 元素数
symbol_set # 元素种类;tuple;会进行排序
types_of_species # 元素种类;Element
formula # 化学式
compsition # 成分;as_dict() 转换成字典形式
cart_coords # 笛卡尔坐标
frac_coords # 分数坐标
lattice # 点阵
.abc # 晶格常数
volume # 体积
density # 密度
center_of_mass # 质心
site_properties # dict;位点性质,如 selective_dynamics
charge # 电荷
# 方法
make_supercell() # 建立超胞
append() # 添加原子位点
remove_species() # 删除元素种类
remove_sites() # 删除原子位点
replace_species() # 替换元素种类
translate_sites() # 移动原子位点
add_site_property() # 添加位点性质
remove_site_property() # 移除位点性质
get_neighbors() # 给定半径,获取给定原子位点的近邻原子
get_all_neighbors() # 给定半径,获取所有原子位点的近邻原子
get_distance() # 获取两个原子位点间的距离
get_space_group_info() # 获取空间群信息
to_cell() # 获取单/原胞
to_conventional() # 获取单胞;调用 to_cell()
to_primitive() # 获取原胞;同上
interpolate() # 在两个构型间插值,用于 NEB 计算
sort() # 排序(原子位点按电负性排序;in place)
get_sorted_structure() # 排序(not in place)
apply_strain() # 对点阵施加应变;默认会修改对象本身
perturb() # 对结构中的原子位点施加随机扰动以破坏对称性
make_supercell() # 构建超胞
# 类方法
from_spacegroup() # 根据空间群构建结构
from_prototype() # 通过原型结构快速构建结构;实际调用的是 from_spacegroup()
# 可支持的 prototype
bcc # Nb
fcc # Al
hcp # Mg
fluorite, caf2 # CaF2
antifluorite # Na2O
rocksalt # NaCl
cscl # CsCl
diamond # Si
zincblende # ZnS
perovskite # BaTiO3
# 获取 Structure 某种元素的原子位点序号
indices_Nb = [i for i, site in enumerate(structure) if site.species_string == "Nb"]
composition¶
- 成分/化学式模块
from pymatgen.core.composition import Composition
comp = Composition("LiFePO4")
# 属性
num_atoms # 原子数
weight # 总的相对原子质量
alphabetical_formula # 化学式,元素按字母顺序排序
reduced_formula # 约化化学式
chemical_system # 所含的化学元素;dict
chemical_system_set # 所含的化学元素;以 - 连接的 str
fractional_composition # 分数成分
reduced_composition # 约化成分
is_element # 是否为纯元素
# 方法
get_atomic_fraction() # 原子百分比
get_wt_fraction() # 质量百分比
get_el_amt_dict() # 返回元素及对应的数目;dict
as_dict() # 基本同上
to_html_string() # 将化学式生成 HTML 格式(htmlify())
to_latex_string() # 将化学式生成 LaTeX 格式(latexify())
to_unicode_string() # 将化学式生成 unicode 格式(unicodeify())
periodic_table¶
- 元素周期表模块,查看元素信息;Element 类继承自 ElementBase
from pymatgen.core.periodic_table import Element
# 属性
data # 所有数据
Z # 原子序数
number # 同上
symbol # 元素符号
long_name # 元素的长名称(单词)
atomic_mass # 相对原子质量
atomic_radius_calculated # 计算的原子半径;经验值
van_der_waals_radius # 范德华半径;经验值
ionic_radii # 离子半径
electronic_structure # 电子结构(可查看元素价电子排布)
n_electrons # 电子数
full_electronic_structure # 完整的电子结构
melting_point # 熔点
boiling_point # 沸点
liquid_range # 液相范围
bulk_modulus # 体莫量
youngs_modulus # 杨氏模量
ionization_energies # 电离能
is_metal # 是否为金属
is_transition_metal # 是否为过渡族金属
# 方法
# 静态方法
print_periodic_table() # 打印元素周期表
sites¶
- 原子位点
lattice¶
- 点阵
from pymatgen.core.lattice import Lattice
# lattice 构建
Lattice([[5, 0, 0], [0, 5, 0], [0, 0, 5]])
Lattice.from_parameters(5, 5, 5, 90, 90, 90)
Lattice.cubic(5)
# 属性
abc # 点阵常数(长度)
angles # 点阵常数(夹角)
a # 点阵常数 a
b # 点阵常数 b
c # 点阵常数 c
alpha # 点阵常数 alpha
beta # 点阵常数 beta
gamma # 点阵常数 gamma
reciprocal_lattice # 倒易点阵
is_orthogonal # 是否为正交胞
is_3d_periodic # 3 个方向是否都是周期性的
matrix # 矩阵
inv_matrix # 逆矩阵
# 方法
is_hexagonal # 是否为六方胞
d_hkl() # 获取晶面间距(不准确,不推荐用)
get_wigner_seitz_cell() # wigner seitz 原胞
get_brillouin_zone() # 布里渊区;倒易点阵的 wigner seitz 原胞
units¶
- pymatgen 相关工具
from pymatgen.util.string import ...
# 函数
formula_double_format() # 将 Li1.0Fe1.0P1.0O4.0 -> LiFePO4
latexify() # 将化学式生成 LaTeX 格式
htmlify() # 将化学式生成 HTML 格式
unicodeify() # 将化学式生成 unicode 格式
latexify_spacegroup() # 将空间群生成 LaTeX 格式
unicodeify_spacegroup() # 将空间群生成 unicode 格式
from pymatgen.util.plotting import ...
# 函数
pretty_plot()
pretty_polyfit_plot()
periodic_table_heatmap() #
format_formula()
get_ax_fig()
add_fig_kwargs() # 装饰器
from pymatgen.util.coord import ...
# 函数
get_angle() # 获取两个向量的夹角
bonds¶
- 键
surface¶
-
表面 slab 模块;总体而言,该模块生成表面模型没有 ASE 相关模块或 atomsk 或 latgen 好用
from pymatge.core.surface import ...
# 方法
# 获取指定晶面指数中的最大数值下其对称性非等同的所有晶面指数
get_symmetrically_distinct_miller_indices()
# 获取指定晶面指数下其对称性等同的所有晶面指数
get_symmetrically_equivalent_miller_indices()
get_d() # 获取 slab 的原子层间距
SlabGenerator类相关属性和方法:
from pymatge.core.surface import SlabGenerator
SlabGenerator # 类;构建指定晶面的 slab 模型
# 参数
initial_structure # 初始结构,需是单胞
miller_index # 晶面密勒指数
min_slab_size # slab 最小尺寸
min_vacuum_size # 真空层最小厚度
center_slab # 是否将原子移至 z 方向中间
in_unit_planes # 设置 min_slab_size 和 min_vacuum_size 参数的单位,False 为 Angstrom,True 为 slab 层数
primitive # 是否将生成的 slab reduce 成 primitive cell
max_normal_search #
reorient_lattice # 使 c 方向平行于第三个点阵矢量
# 方法
get_slabs() # 获取所有的 slab 构型(数量含义为该 slab 模型下不同终端的数量)
# 示例
structure = Structure.from_prototype("bcc", ["Nb"], a=3.32)
slabgen = SlabGenerator(
initial_structure=structure,
miller_index=(1, 1, 0),
min_slab_size=10,
min_vacuum_size=10,
center_slab=True,
in_unit_planes=False,
primitive=True,
)
slabs = slabgen.get_slabs()
slabs[0]
pymatgen.io.ase¶
AseAtomsAdaptor:将 ASE 中的Atoms类与 pymatgen 中的Structure类互相转换
from pymatgen.io.ase import AseAtomsAdaptor
# 静态方法
get_structure() # atoms 转 Structure
get_atoms() # Structure 转 atoms
pymatgen.io.atat¶
- 只有 Mcsqs 类(功能较一般)
pymatgen.io.vasp.help¶
- 查看 VASP 参数 help
from pymatgen.io.vasp.help import VaspDoc
# 静态方法
VaspDoc.get_incar_tags()
VaspDoc.get_help("IBRION")
# 实例方法(需初始化)
VaspDoc().print_help("IBRION")
# 展示 HTML 格式内容
VaspDoc().print_jupyter_help("IBRION")
pymatgen.io.vasp.inputs¶
-
VASP 输入文件模块
-
四种类
Incar、Poscar、Kpoints、Potcar;都有from_dict()、from_file()、write_file()类方法
Incar¶
- 在解析 INCAR 文件时,得到的字典的键和值都是字符串,需要对 INCAR 中不同参数的键的值的类型进行正确的转换,因此定义了
proc_val()函数
from pymatgen.io.vasp.inputs import Incar
Incar(params: dict[str, Any] | None = None)
# 生成 INCAR文件示例
incar_parameters = dict(
ISTART=0,
ISPIN=2,
...
)
incar = Incar.from_dict(incar_parameters)
incar.write_file("INCAR")
Kpoints¶
-
生成 K 点密度的 classmethod 大多都有
force_gamma参数 -
automatic_density_by_vol()类方法生成的三个方向的 K 点密度公式可理解为: reciprocal_density * (2*π / lattice_constant);kppvol参数值设置可参考MPStaticSet类中的代码
from pymatgen.io.vasp.inputs import Kpoints
# 类方法
automatic() # 弃用,建议使用 INCAR 中的 KSPAING 参数
gamma_automatic() # kpts 参数:三个方向的 K 点密度
monkhorst_automatic() # 同上
automatic_density() # 当结构为面心或六方,或最终的 K 点数值为奇数时,会强制使用 Gamma centered meshes
automatic_gamma_density() # 同上;只用 Gamma centered meshes
automatic_density_by_vol() # 算法和 automatic_density() 相同
automatic_density_by_lengths() # 根据晶格常数生成 K 点密度
automatic_linemode() # 计算能带用
# KPOINTS dict
kpoints_dict = {
"comment": "Automatic mesh",
"nkpoints": 0,
"generation_style": "Gamma",
"kpoints": [[10, 10, 10]],
"usershift": [0, 0, 0],
}
# KPOINTS 完整 dict
kpoints_dict = {
'@module': 'pymatgen.io.vasp.inputs',
'@class': 'Kpoints',
'comment': 'k-point density of [20, 20, 20]/[a, b, c]',
'nkpoints': 0,
'generation_style': 'Gamma',
'kpoints': [(4, 4, 2)],
'usershift': (0, 0, 0),
'kpts_weights': None,
'coord_type': None,
'labels': None,
'tet_number': 0,
'tet_weight': 0,
'tet_connections': None,
}
kpoints = Kpoints.from_dict(kpoints_dict)
# 生成 K-Path
from pymatgen.core.structure import Structure
from pymatgen.io.vasp.inputs import Kpoints
from pymatgen.symmetry.bandstructure import HighSymmKpath
structure = Structure.from_file("POSCAR")
kpath = HighSymmKpath(structure)
# division = 20 vaspkit 默认值
kpoints = Kpoints.automatic_linemode(divisions=20, ibz=kpath)
print(kpoints)
Poscar¶
from pymatgen.io.vasp.inputs import Poscar
# 属性
structure # 关联的结构
comment # POSCAR 文件开头的 comment string;# 开头会读取不了
natoms # 原子数目
site_symbols # 原子种类
# 方法
Potcar¶
- 读取和写入 POTCAR 文件的 object,由
PotcarSingleobject (单个 POTCAR) 的列表组成
from pymatgen.io.vasp.inputs import Potcar
# 这里的 symbols 是元素赝势符号,而非元素符号
potcar = Potcar(
symbols=["Nb_sv", "Si"],
functional="PBE",
)
potcar.write_file("POTCAR")
pymatgen.io.vasp.sets¶
-
MPRelaxSet、MPStaticSet等类均继承于VaspInputSet,都有write_input()方法(可能会发生 POSCAR 中的元素重新排序的情况) -
MPRelaxSet没有设置 EDIFFG 参数:MPRelaxSet.write_input() no EDIFFG in INCAR - pymatgen - Materials Science Community Discourse -
MPRelaxSet中的赝势选择相比 MIT project 参数,含更多的价电子;K 点网格密度也比其高 50% -
在 MPRelaxSet.yaml 参数设置文件中,KPOINTS 只能写如下几种参数
# 在 VaspInputSet 类的 kpoints 属性中查看
grid_density # Kpoints.automatic_density()
reciprocal_density # KPoints.automatic_density_by_vol()
length # Kpoints.automatic()(不推荐)
line_density # line mode;手动构建,而非调用 Kpoints.automatic_linemode()
added_kpoints # specific k-points to include
zero_weighted_reciprocal_density # a zero weighted uniform mesh
zero_weighted_line_density # a zero weighted line mode mesh
## yaml 文件
# 弛豫计算;MP 默认的输入文件参数设置
# 没有设置 EDIFFG;EDIFF 设置的是 EDIFF_PER_ATOM: 5.0e-05
# 没有 MPStaticSet.yaml
# MPSCANRelaxSet 有设置 EDIFFG
pymatgen/io/vasp/MPRelaxSet.yaml
# INCAR 文件中的 MAGMOM 元素磁矩参数
pymatgen/io/vasp/VASPIncarBase.yaml
# MIT 团队高通量项目的输入文件参数设置
pymatgen/io/vasp/MITRelaxSet.yaml
## 该模块中的类
MPRelaxSet # 弛豫计算;reciprocal_density: 64
MPStaticSet # 静态计算;参数基于 MPRelaxSet
MPMetalRelaxSet # 金属体系的弛豫计算;参数基于 MPRelaxSet;"ISMEAR": 1, "SIGMA": 0.2,"reciprocal_density": 200
MPHSERelaxSet # 和 MPRelaxSet 一样,有添加 HSE 参数和 vdW 纠正
MatPESStaticSet # 生成 potential energy surface(PES) 数据
MPHSEBSSet # HSE 能带结构计算
MPSOCSet # SOC 计算
MVLElasticSet # MVL 表示 Materials Virtual Lab;弹性常数计算
MVLGWSet # GW 计算
MVLSlabSet # 表面 slab 计算
MVLGBSet # 晶界计算
LobsterSet # Lobster 计算
from pymatgen.io.vasp.sets import VaspInputSet
VaspInputSet(...)
# 参数
user_incar_settings # 自定义 INCAR 参数
user_kpoints_settings # 自定义 KPOINTS 参数
user_potcar_settings # 自定义元素赝势
user_potcar_functional # 自定义泛涵
force_gamma # 是否使用 Gamma-centered K 点生成方式
config_dict # config_dict["POTCAR"]["Mg"]
use_structure_charge # 若为 True,会将体系的电荷赋值给 INCAR 中的 NELECT 参数
# 方法
write_input() # 生成 VASP 计算用的 4 个输入文件
# 参数
output_dir=...
potcar_spec=True
# 静态方法
from_prev_calc() # 基于之前的 VASP 计算目录中生成静态计算输入文件
pymatgen.io.vasp.outputs¶
- 读取并解析 VASP 的输出文件
Outcar¶
- Outcar 类能获取的较普适数据的属性和方法较少(主要解析 Vasprun.xml 文件无法获取到的数据)
from pymatgen.io.vasp.ouputs import Outcar
# 属性
drift # 每个离子步的 Total drift
run_stats # "Total CPU time used (sec)" 相关内容
final_energy # "energy(sigma->0)"
final_energy_wo_entrp # "energy without entropy"
final_fr_energy # "free energy TOTEN"
# 方法
read_pattern() # 通用 pattern 解析
read_table_pattern() # 解析类列表数据;分成 header、main body 和 footer 三部分,返回 main body 中的内容
read_neb() # 读取 VASP 中常规的 NEB 或 CINEB 数据
# 示例
outcar = Outcar(...)
# 获取原子位置坐标和受力
table_data = outcar.read_table_pattern(
header_pattern=r"\sPOSITION\s+TOTAL-FORCE \(eV/Angst\)\n\s-+",
row_pattern=r"\s+([+-]?\d+\.\d+)\s+([+-]?\d+\.\d+)\s+([+-]?\d+\.\d+)\s+([+-]?\d+\.\d+)\s+([+-]?\d+\.\d+)\s+([+-]?\d+\.\d+)",
footer_pattern=r"\s--+",
postprocess=lambda x: float(x),
last_one_only=False,
)
Oszicar¶
- Oszicar 类的
final_energy属性选择的是E0;Vasprun 类的final_energy属性选择的也是E0
from pymatgen.io.vasp.ouputs import Oszicar
# 属性
ionic_steps # 离子步数据
electronic_steps # 电子步数据
final_energy # E0
all_energies # 电子步 + 离子步
# 方法
as_dict() # key 为 ionic_steps 和 electronic_steps 的 dict
Vasprun¶
BSVasprun 类:Vasprun 的优化版本(继承至 Vasprun),只解析能带结构的本征值(忽略结构,参数等)
from pymatgen.io.vasp.ouputs import Vasprun
# 属性
converged # 检查离子步、电子步是否都收敛
converged_electronic # 检查电子步是否收敛
converged_ionic # 检查离子步是否收敛
incar # INCAR 文件内容
kpoints # KPOINTS 文件内容
potcar_spec # POTCAR 种类
potcar_symbols # POTCAR 符号
vasp_version # VASP 版本
initial_structure # 初始构型
final_structure # 最终构型
structures # 每个离子步构型
final_energy # 最终能量 E0
nionic_steps # 离子步步数
ionic_steps # 每步离子步内容
complete_dos # 获取 DOS 数据
tdos # 总态密度
idos # 积分态密度
pdos # 类型是列表;索引方式 pdos[atomindex][orbitalindex]
# 方法
as_dict() # 将解析的 vasprun.xml 数据转换为 dict
get_computed_entry() # 将 vasprun.xml 中的计算结果转换为 ComputedEntry 对象
get_band_structure() # 获取能带结构数据;line_mode=True
# 每个 ionic_step 所含的数据 dict key
dict_keys(
[
"e_fr_energy",
"e_wo_entrp",
"e_0_energy",
"forces",
"stress",
"electronic_steps",
"structure",
]
)
- Vasprun 类在解析未收敛的计算时,会有 Warning,并说明电子步和离子步的收敛情况
UnconvergedVASPWarning: vasprun.xml is an unconverged VASP run.
Electronic convergence reached: True.
Ionic convergence reached: False.
warnings.warn(msg, UnconvergedVASPWarning)
Vaspout¶
- 解析 vaspout.h5 文件(pymatgen 2025.03.10 版本支持);需安装 h5py 包
from pymatgen.io.vasp.outputs import Vaspout
vaspout = Vaspout("vaspout.h5")
# 属性
incar
poscar
kpoints
potcar
converged
initial_structure
final_structure
final_energy
其他¶
pymatgen.io.lammps¶
data¶
- 解析 LAMMPS data 格式文件
from pymatgen.io.lammps.data import LammpsData
# 方法
write_file() # 写入 LAMMPS data 格式文件
# 类方法
from_structure() # 从 Structure 导入
from_file() # 从文件导入
# 属性
# 以 Pandas DataFrame 格式存储信息
atoms # 原子信息(type x y z 等)
box # 盒子信息
masses # 相对原子质量
atom_style # atom_style 类型
velocities # 速度
force_field # 力场信息
topology # 拓扑信息
pymatgen.electronic_structure¶
-
电子结构相关工具与分析(能带和态密度);可绘制能带、DOS、能带 + DOS
-
Plotter 类的 sigma 参数使绘制出的图平滑(sigma=0.05 相对好一些;若平滑后使原本的部分数据信息损失,建议不设置该参数!)
态密度¶
-
态密度 DOS 数据获取与绘图
- label 颜色自定义:修改源代码(获取 ax 的方法不行)
- 使用 pymatgen 模块绘制 DOS 图时,相对会耗时一些,建议将 DOS 数据获取后单独存储为数据文件自己绘制
import matplotlib.pyplot as plt
from pymatgen.electronic_structure.plotter import DosPlotter
from pymatgen.io.vasp.outputs import Vasprun, BSVasprun
from pymatgen.electronic_structure.core import OrbitalType, Orbital, Spin
from pymatgen.core.periodic_table import Element
# 获取态密度数据
dos_vasprun=Vasprun("./dos/vasprun.xml")
dos_data=dos_vasprun.complete_dos
# 获取费米能级
fermi = dos_data.efermi
# 整体能量平移
energy = dos_data.energies - fermi
dos_data.densities # 体系总态密度
dos_data.get_spd_dos() # 体系分态密度
dos_data.get_element_dos() # 元素总态密度
dos_data.get_element_spd_dos() # 元素分态密度
dos_data.get_site_dos() # 原子总态密度
dos_data.get_site_spd_dos() # 原子分态密度
# 方式 1
# 体系总态密度数据
total_densities = dos_data.densities
total_densities[Spin.up]
# 体系分态密度数据
spd_dos = dos_data.get_spd_dos()
dos_s = spd_dos[OrbitalType.s].densities[Spin.up]
# 元素总态密度数据
element_dos = dos_data.get_element_dos()
dos_Si = element_dos[Element("Si")].densities[Spin.up]
# 方式 2
# 提取 TDOS 数据
dos_vasprun.tdos.as_dict()["energies"]
dos_vasprun.tdos.as_dict()["densities"]["1"]
# 提取原子位点 PDOS 数据
dos_vasprun.pdos[0][Orbital.s][Spin.up]
# 态密度(总)绘制
dos_plot = DosPlotter()
dos_plot.add_dos("Total", dos=dos_data)
dos_plot.get_plot()
# 态密度(投影到 spd 轨道)绘制
pdos_plot = DosPlotter()
pdos_plot.add_dos_dict(dos_data.get_spd_dos())
pdos_plot.get_plot()
# 态密度(投影到元素)绘制
edos_plot = DosPlotter()
edos_plot.add_dos_dict(dos_data.get_element_dos())
edos_plot.get_plot()
# 态密度(投影到元素 spd 轨道)绘制
edos_plot = DosPlotter()
pdos_Nb = dos_data.get_element_spd_dos("Nb")
dos_plot.add_dos("Nb(s)", dos=pdos_Nb[OrbitalType.s])
dos_plot.add_dos("Nb(p)", dos=pdos_Nb[OrbitalType.p])
dos_plot.add_dos("Nb(d)", dos=pdos_Nb[OrbitalType.d])
edos_plot.get_plot()
# 态密度(投影到原子)绘制
ados_plot = DosPlotter()
ados_plot.add_dos("Site 0 Total", dos=dos_data.get_site_dos(structure[0]))
ados_plot.get_plot()
# 态密度(投影到原子 spd 轨道)绘制
apdos_plot = DosPlotter()
dos_site0 = dos_data.get_site_spd_dos(structure[0])
apdos_plot.add_dos("Nb0(s)", dos=dos_site0[OrbitalType.s])
apdos_plot.add_dos("Nb0(p)", dos=dos_site0[OrbitalType.p])
apdos_plot.add_dos("Nb0(d)", dos=dos_site0[OrbitalType.d])
apdos_plot.get_plot()
# 对 get_plot() 返回的 Axes 对象进行进一步的操作
ax = dos_plot.get_plot()
ax.set_xlim(...)
plt.savefig(...) # 保存图片,可不使用 dos_plot.save_plot()
能带¶
import matplotlib.pyplot as plt
from pymatgen.electronic_structure.core import OrbitalType, Orbital
from pymatgen.io.vasp.outputs import Vasprun, BSVasprun
from pymatgen.electronic_structure.plotter import (
BSPlotter,
BSPlotterProjected,
BSDOSPlotter,
)
# 获取能带数据
bs_vasprun = Vasprun("./bs/vasprun.xml", parse_projected_eigen=True)
bs_data = bs_vasprun.get_band_structure(line_mode=True)
# 能带绘制
bs_plot = BSPlotter(bs=bs_data)
bs_plot.get_plot()
# 能带 + 态密度绘制
bsdos_plot = BSDOSPlotter(bs_projection=None, dos_projection=None)
bsdos_plot.get_plot(bs=bs_data, dos=dos_data)
COHP¶
- COHP (Crystal orbital Hamilton population) 与 ICOHP 之间的区别是什么
sampleScript/RHEAs/COHP.py at master · Jeffery-Li0513/sampleScript · GitHub
from pymatgen.electronic_structure.cohp import Cohp, get_integrated_cohp_in_energy_range
from pymatgen.electronic_structure.plotter import CohpPlotter
from pymatgen.io.lobster import Cohpcar, Icohplist
pymatgen.analysis¶
structure_matcher¶
- 查看两个结构之间的相似度(是否相同/接近)
from pymatgen.analysis.structure_matcher import StructureMatcher
sm = StructureMatcher()
sm.fit(structure1, structure2)
# 参数
ltol # 默认值 0.2
stol # 默认值 0.3
angle_tol # 默认值 5
eos¶
- EOS 类:
BirchMurnaghan、Birch、Murnaghan、PourierTarantola、Vinet等
from pymatgen.analysis.eos import BirchMurnaghan
eos = BirchMurnaghan(volumes=..., energies=...)
eos.fit() # EOS 拟合
eos.results # EOS 拟合结果
eos.v0 # 平衡体积拟合值
eos.e0 # 平衡能量拟合值
eos.b0_GPa # 体模量 B 拟合值
eos.plot() # 绘制 EOS 拟合曲线
interface¶
-
Working with Surfaces and Interfaces - The Materials Project Workshop
-
pymatgen 界面模型生成无法指定两种 slab 的层数:https://matsci.org/t/building-a-slab-and-interface/45317
-
生成所有可能匹配的超晶格的过程:
- 减少表面/界面(surfaces)晶格向量并计算表面/界面的面积
- 在最大允许面积内生成所有超晶格变换
- 对于每个超晶格集:
- 减少超晶格矢量
- 检查 film 和 substrate 表面超晶格之间的长度和角度
from pymatgen.analysis.interfaces.coherent_interfaces import CoherentInterfaceBuilder
from pymatgen.analysis.interfaces.zsl import ZSLGenerator
zsl # Zur 和 McGill 晶格匹配算法 模块
ZSLGenerator # 基于 zsl 算法的界面生成 类
coherent_interfaces #
CoherentInterfaceBuilder # 共格界面构建 类
elasticity¶
# 施加正应变/剪切应变,生成变形后的结构
from pymatgen.analysis.elasticity import DeformedStructureSet
DeformedStructureSet()
electronic_structure¶
- pymatgen 电子结构相关分析很多都是建立在 vasprun.xml 文件中提取数据之上的(与 vaspkit 有不同)
phase_diagram¶
-
可绘制二、三、四元的相图
-
无法直接使用
pymatgen.entries中的Entry类初始化,会报错,energy参数为ABC抽象类型(Doc 有提及),而是用pymatgen.analysis.phase_diagram中的 PDEntry 类初始化 -
label 字体大小无法调节:How control fontsize in PDPlotter? - pymatgen - Materials Science Community Discourse
# 绘制相图 Convex Hull
from pymatgen.analysis.phase_diagram import PDEntry, PhaseDiagram
from pymatgen.core.composition import Composition
entry1 = PDEntry(composition=..., energy=...)
entry2 = ...
...
entries = [entry1, entry2, ...]
phasediagram = PhaseDiagram(entries) # 稳定相
# 属性
stable_entries # 稳定相及其对应能量
# 方法
get_decomposition() # 获取特定构型成分分解成哪些稳定相及其比例
get_decomp_and_e_above_hull() #
get_plot() # 绘制相图
# label 字体大小无法修改 可能会导致有重叠
ax = phasediagram.get_plot(
backend="matplotlib", # 绘图后端;ploty 或 matplotlib
show_unstable=False, # 是否显示非稳定构型
# label_stable=False, # 是否显示 label
)
ax.figure.savefig()
diffraction¶
- XRD 绘制:
from pymatgen.analysis.diffraction.xrd import XRDCalculator
from pymatgen.analysis.diffraction.neutron import NDCalculator
from pymatgen.analysis.diffraction.tem import TEMCalculator
c = XRDCalculator()
# 方法
get_plot(structure) # 绘制结构的 XRD
get_pattern(structure) # 获取衍射花样
get_pattern(structure).hkls
get_pattern(structure).d_hkls
adsorption¶
from pymatgen.analysis.adsorption import AdsorbateSiteFinder, plot_slab
# 方法
find_adsorption_sites() # 搜索吸附位点
add_adsorbate() # 添加吸附原子/分子
# 函数
plot_slab() # 可视化吸附位点
prototype¶
- 匹配晶体结构的 prototype(调用的是 AFLOW 的 prototype 数据)
diffusion¶
- 使用
# 使用 IDPP 方法进行插值
from pymatgen.analysis.diffusion.neb.pathfinder import IDPPSolver
# 方法
run()
# 静态方法
from_endpoints()
idpp_solver = IDPPSolver.from_endpoints()
idpp_solver.run()
# 概率密度
from pymatgen.analysis.diffusion.aimd.pathway import ProbabilityDensityAnalysis
from pymatgen.analysis.diffusion.analyzer import DiffusionAnalyzer
diffusion_ana = DiffusionAnalyzer.from_structures()
diffusion_ana.export_msdt()
diffusion_ana.conductivity
pda = ProbabilityDensityAnalysis.from_diffusion_analyzer(diffusion_ana)
pda.to_chgcar()
pymatgen.symmetry.bandstructure¶
- 寻找高对称 K 点
from pymatgen.symmetry.bandstructure import HighSymmKpath
structure = ...
kpath = HighSymmKpath(structure=structure)
kpath.kpath
kpath.kpath["kpoints"]
kpath.kpath["path"]
pymatgen.symmetry.analyzer¶
SpacegroupAnalyzer¶
- 空间群分析
from pymatgen.symmetry.analyzer import SpacegroupAnalyzer
sga_analyzer = SpacegroupAnalyzer(structure, symprec=...,)
# 默认对称性精度为 0.01
# 弛豫后的结构,精度可适当放宽(0.1 为 MP 使用的精度)
# 方法
get_conventional_standard_structure() # 获取单胞
get_primitive_standard_structure() # 获取原胞
get_symmetrized_structure() # 获取对称性结构
get_symmetry_dataset() # 获取结构的对称性数据集
get_crystal_system() # 获取晶系(源码含空间群与晶系之间的关系)
get_space_group_number() # 空间群编号(编号越小,对称性越低)
get_space_group_symbol() # 空间群符号
get_crystal_system() # 获取晶系
symmetry_dataset = sga_analyzer.get_symmetry_dataset()
# dataset 中的信息
number # 空间群序号
international # 空间群 International symbol
equivalent_atoms # 等同原子
wyckoffs # Wyckoff letters
pymatgen.transformations¶
-
参考:
-
简单的变换操作:添加和删除原子位点,元素替换,到更高级的一对多的转换
-
pymatgen 无置换 Wyckoff Site 中的元素的类
-
SQSTransformation类中调用的是 ATAT 中的 mcsqs 工具或 ICET 中的 enumeration、monte carlo 模块;建议直接使用其调用的原生工具
from pymatgen.transformations.standard_transformations import ...
from pymatgen.transformations.advanced_transformations import ...
from pymatgen.transformations.site_transformations import ...
# 使用方法
structure = ...
transformation = XXX()
structure_new = transformation.apply_transformation(structure)
# standard_transformations 中的类
RemoveSpecieTransformations # 删除元素种类
AutoOxiStateDecorationTransformation # 自动平衡结构中的价态信息
ChargedCellTransformation # 给结构施加额外电荷,使体系整体带正电或者负电
SubstitutionTransformation # 元素替换;参数 species_map
# advanced_transformations 中的类
SQSTransformation # SQS 结构
EnumerateStructureTransformation # 枚举结构
# site_transformations 中的类
ReplaceSiteSpeciesTransformation # 原子位点元素种类替换
# 以上类都有的方法
apply_transformation(structure) # 施加变换操作到构型上
# 示例
# 获取 sqs 结构
sqs = SQSTransformation([2, 2, 2])
# 枚举无序结构
# reference: https://github.com/luzihen/pymatgen_examples/blob/master/enumerate_ordering.py
enum = EnumerateStructureTransformation()
enumerated = enum.apply_transformation(structure, return_ranked_list=100) # return no more than 100 structures
# 元素置换
species_map = {"Si": "C"}
# 分数占据
species_map = {"Si":{"Si":0.9, "C":0.1}}
pymatgen.phonon¶
API¶
-
调用 MP API 获取 MP 数据
-
参考:
-
MPRester through proxy server - pymatgen - Materials Science Community Discourse
-
新 API:
-
安装
- 新 API 使用
# 新 API 模块导入
from mp_api.client import MPRester
with MPRester("api-key") as mpr:
docs = mpr.materials.summary.search(...)
# 查看可获取内容的字段,可用做筛选 query data 的参数
mpr.materials.summary.available_fields
# 根据元素获取 ComputedStructureEntry
mpr.get_entries_in_chemsys()
# 能带、DOS
mpr.get_bandstructure_by_material_id()
mpr.get_dos_by_material_id()
# 声子
mpr.get_phonon_bandstructure_by_material_id()
mpr.get_phonon_dos_by_material_id()
# 相图(可绘制二、三、四元相图)
mpr.materials.thermo.get_phase_diagram_from_chemsys(chemsys=...,)
chemsys="Ti-Al" # 二元
chemsys="Ti-Al-Nb" # 三元
chemsys="Ti-Al-Nb-Zr" # 四元
# search() 参数
material_ids=["mp-149"] # 根据材料 ID
chemsys="Si-O", # 仅含 Si O 两种元素的材料
elements=["Si", "O"] # 至少含 Si O 两种元素的材料
has_props=["dos"] # 是否有该性质
fields=["band_gap"] # 字段
is_stable=True # 是否为稳定材料
# fields 参数常用字段
material_id # MP 对该材料标注的 ID;需 str()
composition_reduced # 成分(约化);需 as_dict()
formula_pretty # 化学式(约化)
structure # 结构
symmetry
.crystal_system # 晶系;需 str()
.symbol # 空间群
nsites # 构型原子数
energy_per_atom # 能量/原子
formation_energy_per_atom # 形成能/原子
energy_above_hull # 形成能与在 Hull 上的形成能差值
is_stable # 材料是否是稳定的
fields_not_requested # 列出未请求的字段
- 旧 API 使用
# 旧 API模块导入
from pymatgen.ext.matproj import MPRester
# 从 MP 获取结构
with MPRester("api-key") as mpr:
# 相关方法源码路径 pymatgen/ext/matproj_legacy.py
mpr.query() # 采用类 MongoDB 的 query 语法从 MP 中获取数据
mpr.get_structure_by_material_id() # 根据材料 ID 获取结构
mpr.get_download_info() # 获取来自 NoMaD repository 的裸 VASP 输出文件 URL
mpr.get_gb_data() # 获取晶界数据
mpr.get_surface_data() # 获取表面能数据
# query 参数
criteria # query 准则
properties # 性质
# properties 列表内容
formula
spacegroup
formation_energy_per_atom
elasticity
获取表面性质、晶界数据¶
-
只能通过 API 下载表面性质、晶界构型及对应数据(对应于页面 Properties 部分的 Surface、Heterostructures 内容)
-
获取表面性质、晶界数据
import os from mp_api.client import MPRester API_KEY = os.getenv("PMG_MAPI_KEY") with MPRester(api_key=API_KEY) as mpr: # 获取 表面性质 数据 surface_properties_doc = mpr.materials.surface_properties.search(material_ids=["mp-75"]) print(len(surface_properties_doc)) # 1 print(len(surface_properties_doc[0].surfaces)) # 13 print(surface_properties_doc[0].surfaces[0].miller_index) print(surface_properties_doc[0].surfaces[0].structure) # 获取 晶界 数据 grain_boundary_doc = mpr.materials.grain_boundaries.search(material_ids=["mp-75"]) print(len(grain_boundary_doc)) # 10 print(grain_boundary_doc[0].sigma) print(grain_boundary_doc[0].initial_structure) print(grain_boundary_doc[0].final_structure) -
查看表面性质、晶界可获取的数据
from emmet.core.grain_boundary import GrainBoundaryDoc
from emmet.core.surface_properties import SurfacePropDoc
print(GrainBoundaryDoc.model_fields.keys())
dict_keys(
[
"material_id",
"sigma",
"type",
"rotation_axis",
"gb_plane",
"rotation_angle",
"gb_energy",
"initial_structure",
"final_structure",
"pretty_formula",
"w_sep",
"cif",
"chemsys",
"last_updated",
]
)
print(SurfacePropDoc.model_fields.keys())
dict_keys(
[
"surfaces",
"weighted_surface_energy_EV_PER_ANG2",
"weighted_surface_energy",
"surface_anisotropy",
"pretty_formula",
"shape_factor",
"weighted_work_function",
"has_reconstructed",
"material_id",
"structure",
]
)