ACE 学习与使用¶
约 1584 个字 249 行代码 11 张图片 预计阅读时间 8 分钟
介绍¶
WIP...
参考资料¶
WIP...
相关问题¶
CE 与 ACE 之间的区别(本质上没区别,后者拟合的参数更多)
目前暂无 CE 与 ACE 模型之间的对比
ACE 与 MTP 在数学上没有差别
ACE 模型接入到 LAMMPS,其运行效率如何 待测试 基函数超过 10000,计算效率可能会降低
ACE 目前有无 MC 的接口(非 MD 的 MC) 没有
研究不同相的转变温度,训练集需添加所有相的构型?
公式的变换,考虑了所有的排列甚至虚拟的团簇(引入了非物理的自相互作用项),转换成了张量积的结构,可以利用这种结构构建一个高效的评估方案
\(c\) 参数向量
\(B\) 基函数向量(多体势涉及),具有旋转、镜像、平移对称性
\(V^{(0)}\) 单体项 单个孤立原子的能量
basis function 基函数(含 ridial 和 angular)ridial 和 angular 函数之间互相独立
由 \(\phi\) 变成 \(B\),考虑了其对称性
one-particle 基矢
ridial 基矢 : 它包含了相互作用范围(截止半径,rcut),并可进行调整,以捕捉有关相互作用原子的粗略定性信息。
数据库 随机扰动?
acepotential.jl 相关问题
权重 weight E F V 不是按百分比来的? 理论上可以很大
高度结构化,与构型的原子数成比例
ACE 是关于 c 线性的,最小化最小二乘损失函数
smoothness prior. 通常为 3 或 4 筛除不合理(unphysics)的构型,在参数拟合过程前,与参数拟合没有关系
二体和多体分别保存成文件 转换时会自动生成两个文件
孤立原子的参考态能量 CASTEP 计算得到
参数拟合默认贝叶斯方法
acemodel 与 acebasis 进行模型构建方法之间的区别?
迭代学习、不确定度(超主动学习?)
GitHub - ACEsuit/ACEHAL at 1575e41fc7c6284be7b54f8fa81eac7184656bef
参数估计:ridge 和 bayesian 两大类
非线性 ACE? 作用:降低复杂度 ACE+DNN, GNN → MACE(精度更高,但参数量很大,能达到百万级别)
将 J 转换成 c 的形式,trick,减小计算量
矩阵 A 保证平移和置换对称性,矩阵 B 保证旋转对称性(晶体对称性)
ACE 相关
ACEPotentials.jl 使用¶
参考资料¶
模型测试:python-ase
将 OUTCAR 每个离子步信息转换成 extxyz 格式
Convert VASP OUTCAR to extxyz file for NequIP input · GitHub
安装¶
安装
创建 conda 环境
安装 pyjulip(call julia and evaluate ACE potentials)
创建 ACEPotential Julia 项目环境,添加注册表和 packages
using Pkg
Pkg.activate(".")
# 添加注册表
Pkg.Registry.add("General") # only needed when installing Julia for the first time
Pkg.Registry.add(RegistrySpec(url="https://github.com/ACEsuit/ACEregistry"))
# 添加 packages
Pkg.add("ACEpotentials")
# 在使用 python-ase 时,可能需要在默认 Julia 环境中添加以下 packages
add ASE JuLIP ACE1
master 和 siyuan 上的 github https 连接性不是很好,这步可能需耗时较长或不成功,建议添加国内镜像
相关问题¶
思源相关报错
ERROR: Unable to load dependent library /dssg/home/acct-mseklt/mseklt/yangsl/src/julia-1.9.4/bin/../lib/julia/libjulia-codegen.so.1
Message:/lib64/libstdc++.so.6: version `GLIBCXX_3.4.26' not found (required by /dssg/home/acct-mseklt/mseklt/yangsl/src/julia-1.9.4/bin/../lib/julia/libjulia-codegen.so.1)
原因:gcc 版本低
Traceback (most recent call last):
File "/home/yangsl/scripts/ACE/3-ACEPotential/model_verification.py", line 6, in <module>
import pyjulip
File "/home/yangsl/src/miniconda3/envs/acep/lib/python3.10/site-packages/pyjulip.py", line 7, in <module>
jl = Julia(compiled_modules=False)
File "/home/yangsl/src/miniconda3/envs/acep/lib/python3.10/site-packages/julia/core.py", line 519, in __init__
self._call("const PyCall = Base.require({0})".format(PYCALL_PKGID))
File "/home/yangsl/src/miniconda3/envs/acep/lib/python3.10/site-packages/julia/core.py", line 555, in _call
self.check_exception(src)
File "/home/yangsl/src/miniconda3/envs/acep/lib/python3.10/site-packages/julia/core.py", line 609, in check_exception
raise JuliaError(u'Exception \'{}\' occurred while calling julia code:\n{}'
julia.core.JuliaError: Exception 'ArgumentError' occurred while calling julia code:
const PyCall = Base.require(Base.PkgId(Base.UUID("438e738f-606a-5dbb-bf0a-cddfbfd45ab0"), "PyCall"))
使用 ACEpotentials 势函数¶
ACEpotentials 教程:GitHub - wcwitt/ACEpotentials-Tutorial
python-ase¶
python-ase 示例代码
import pyjulip
from ase.io import read
calc = pyjulip.ACE1("first_potential.json")
ats = read('atoms_object.xyz')
ats.calc = calc
print(ats.get_potential_energy())
python 调用 julia 耗时较长,模型预测耗时只需不足 1s
ACE model: 20231204_285.0.json
configuration: test.xyz
predicted total energy: -26.342406 eV
predicted energy per atom: -4.390401 eV/atom
Total energy error: 0.94292 eV
energy per atom error 0.157153 eV/atom
Time cost: 8.391s
------------------------------
ACE model: 20231204_365.0.json
configuration: test.xyz
predicted total energy: -26.566639 eV
predicted energy per atom: -4.427773 eV/atom
Total energy error: 0.718687 eV
energy per atom error 0.119781 eV/atom
Time cost: 0.319s
------------------------------
ACE model: 20231204Eref_365.0.json
configuration: test.xyz
predicted total energy: -26.560462 eV
predicted energy per atom: -4.426744 eV/atom
Total energy error: 0.724864 eV
energy per atom error 0.12081 eV/atom
Time cost: 0.419s
------------------------------
ACE model: 1204_large_375.5.json
configuration: test.xyz
predicted total energy: -26.425927 eV
predicted energy per atom: -4.404321 eV/atom
Total energy error: 0.859399 eV
energy per atom error 0.143233 eV/atom
Time cost: 0.341s
------------------------------
ACE model: 20231204_285.0.json
configuration: test_2.xyz
predicted total energy: -45.062008 eV
predicted energy per atom: -7.510335 eV/atom
Total energy error: 0.140886 eV
energy per atom error 0.023481 eV/atom
Time cost: 0.18s
------------------------------
ACE model: 20231204_365.0.json
configuration: test_2.xyz
predicted total energy: -45.042835 eV
predicted energy per atom: -7.507139 eV/atom
Total energy error: 0.160059 eV
energy per atom error 0.026677 eV/atom
Time cost: 0.144s
------------------------------
ACE model: 20231204Eref_365.0.json
configuration: test_2.xyz
predicted total energy: -45.100551 eV
predicted energy per atom: -7.516759 eV/atom
Total energy error: 0.102343 eV
energy per atom error 0.017057 eV/atom
Time cost: 0.348s
------------------------------
ACE model: 1204_large_375.5.json
configuration: test_2.xyz
predicted total energy: -45.065638 eV
predicted energy per atom: -7.51094 eV/atom
Total energy error: 0.137256 eV
energy per atom error 0.022876 eV/atom
Time cost: 0.434s
------------------------------
ACE model: 1209_375.0.json
configuration: test.xyz
predicted total energy: -26.472107 eV
predicted energy per atom: -4.412018 eV/atom
Total energy error: 0.813219 eV
energy per atom error 0.135536 eV/atom
Time cost: 6.503s
------------------------------
ACE model: 1209_375.0.json
configuration: test_2.xyz
predicted total energy: -44.836956 eV
predicted energy per atom: -7.472826 eV/atom
Total energy error: 0.365938 eV
energy per atom error 0.06099 eV/atom
Time cost: 0.283s
------------------------------
ACE model: 1213_BLR_387.0.json
configuration: test.xyz
predicted total energy: -26.574053 eV
predicted energy per atom: -4.429009 eV/atom
Total energy error: 0.711273 eV
energy per atom error 0.118545 eV/atom
Time cost: 6.556s
------------------------------
ACE model: 1213_BLR_387.0.json
configuration: test_2.xyz
predicted total energy: -44.748131 eV
predicted energy per atom: -7.458022 eV/atom
Total energy error: 0.454763 eV
energy per atom error 0.075794 eV/atom
Time cost: 0.554s
------------------------------
PACE-LAMMPS 接口¶
在超算上安装 ML-PACE package 的 LAMMPS
# 导入 oneAPI 套件
module purge
module load intel-oneapi-compilers/2021.4.0
module load intel-oneapi-mkl/2021.4.0
module load intel-oneapi-mpi/2021.4.0
module load intel-oneapi-tbb/2021.4.0
# git clone -b release https://github.com/lammps/lammps
# cd lammps
mkdir build-pace && cd build-pace
wget -O libpace.tar.gz https://github.com/wcwitt/lammps-user-pace/archive/main.tar.gz
cmake \
-C ../cmake/presets/most.cmake \
-C ../cmake/presets/oneapi.cmake \
-D PKG_ML-PACE=yes \
-D PACELIB_MD5=$(md5sum libpace.tar.gz | awk '{print $1}') \
../cmake
make
LAMMPS 中 in 文件 pair_coeff 参数中的元素顺序需与构型文件、yace 文件中的元素顺序一致
N 数值见 .table 文件
pair_style hybrid/overlay pace table spline <N>
pair_coeff * * pace potential.yace I Cs Pb
pair_coeff 1 1 table potential_pairpot.table I_I
pair_coeff 1 2 table potential_pairpot.table I_Cs
pair_coeff 1 3 table potential_pairpot.table I_Pb
pair_coeff 2 2 table potential_pairpot.table Cs_Cs
pair_coeff 2 3 table potential_pairpot.table Cs_Pb
pair_coeff 3 3 table potential_pairpot.table Pb_Pb
LAMMPS MC 测试
4 核
Loop time of 4089.68 on 4 procs for 40000 steps with 2000 atoms
Performance: 0.845 ns/day, 28.401 hours/ns, 9.781 timesteps/s, 19.561 katom-step/s
99.6% CPU use with 4 MPI tasks x 1 OpenMP threads
8 核
Loop time of 1551.08 on 8 procs for 20000 steps with 2000 atoms
Performance: 1.114 ns/day, 21.543 hours/ns, 12.894 timesteps/s, 25.789 katom-step/s
99.7% CPU use with 8 MPI tasks x 1 OpenMP threads
MACE 相关 projects¶
MACE 相关:GitHub - jharrymoore/ml_fep
GitHub - jharrymoore/MACE-OFF23-SC
GitHub - YangshuaiWang/FoundationModel0
MACE LAMMPS 插件:GitHub - XJTU-ICP/mace_lammps_plugin
非线性 ACE:GitHub - ICAMS/python-ace
GitHub - RokasEl/mace-mp-umap: UMAP analysis using a pretrained MACE model
MACE LAMMPS 接口编译:MACE in LAMMPS — mace 0.1.0 documentation
mace-mp 报错(GPU 资源不足;对)
MACE-MP 模型
# small
2023-12-10-mace-128-L0_energy_epoch-249.model
# medium
2023-12-03-mace-128-L1_epoch-199.model
# large
MACE_MPtrj_2022.9.model
cace 相关:GitHub - BingqingCheng/cacefit
Christoph Ortner 教授:cortner (Christoph Ortner) · GitHub
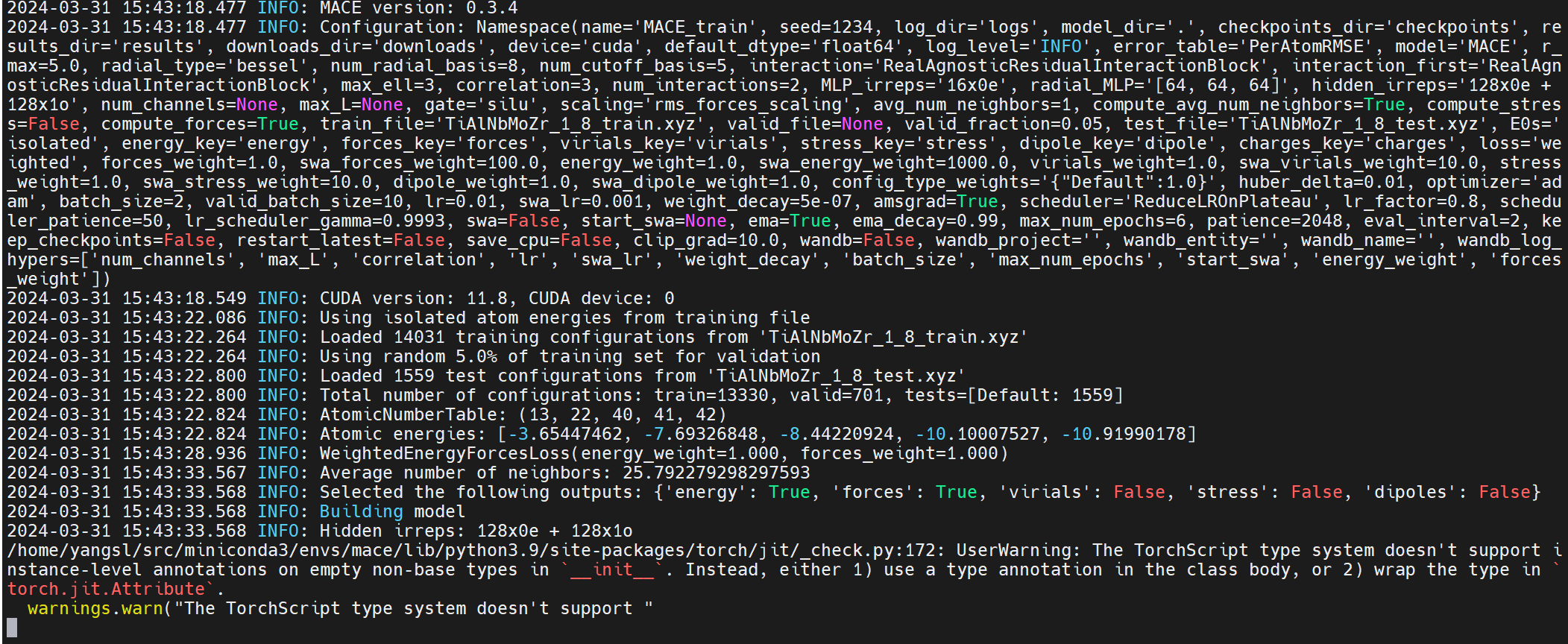
需要 2G 左右显存
CUDA_VISIBLE_DEVICES=1 \
mace_run_train \
--name="MACE_train" \
--train_file="TiAlNbMoZr_1_8_train.xyz" \
--valid_fraction=0.05 \
--test_file="TiAlNbMoZr_1_8_test.xyz" \
--energy_weight=1.0 \
--forces_weight=1.0 \
--E0s="{13: -0.057722998, 22: -1.1935167, 40: -1.3186995, 41: -0.63860806, 42: -0.22478502}" \
--lr=0.01 \
--scaling="rms_forces_scaling" \
--batch_size=2 \
--max_num_epochs=6 \
--ema \
--ema_decay=0.99 \
--amsgrad \
--default_dtype="float64" \
--device=cuda \
--restart_latest \
--seed=1234
# E0s 参数正确写法 原子序数
# 13 Al; 22 Ti; 40 Zr; 41 Nb; 42 Mo
--E0s="{13: -0.057722998, 22: -1.1935167, 40: -1.3186995, 41: -0.63860806, 42: -0.22478502}"
--E0s="isolated"
# E0s 参数错误写法
--E0s="{'Ti': -1.1935167, 'Al': -0.057722998, 'Nb': -0.63860806, 'Mo': -0.22478502, 'Zr': -1.3186995}"
--lr # optimizer 的学习率
--ema # 使用 Exponential Moving Average
--swa # use Stochastic Weight Averaging, which decreases the learning rate and increases the energy weight at the end of the training to help converge them
--amsgrad # 使用 optimizer 的 amsgrad variant
--scaling # {std_scaling,rms_forces_scaling,no_scaling} type of scaling to the output
--restart_latest # restart optimizer from latest checkpoint
在深度学习中,SWA(Stochastic Weight Averaging)是一种提高模型泛化能力的技术。它的核心思想是在训练过程中的不同时间点收集模型的权重,然后计算这些权重的平均值,以此作为最终模型的权重。这种方法可以被视作一种集成学习的技术,因为它综合了训练过程中多个模型的知识。
Exponential Moving Average (EMA) 是一种在时间序列数据分析中广泛使用的技术,特别是在金融分析和信号处理中。它也被应用于机器学习领域,尤其是在训练深度神经网络时用于平滑模型参数。EMA 通过给予更近期数据点更高的权重而不是平均分配权重来计算平均值,与简单移动平均 (Simple Moving Average, SMA) 相比,EMA 能更快地反映数据的最新变化趋势。
-
Batch Size:指的是在训练神经网络时,每次传递给网络并用于计算梯度更新的数据样本的数量。简单来说,如果你有 1000 个训练样本,你的 batch size 是 100,那么这意味着你的神经网络每次会接收 100 个数据样本进行学习,并基于这 100 个样本更新一次网络的权重。使用较小的 batch size 可以提高模型训练过程中的内存效率,并且可以增加模型更新的频率,有助于模型更快地收敛。但是,太小的 batch size 可能会导致训练过程变得不稳定。另一方面,较大的 batch size 可以提高数据处理的效率,使训练过程更稳定,但可能需要更多的内存,并且可能导致模型在找到全局最小值方面的性能不如较小的 batch size。
-
Epochs:指的是将整个训练数据集完整地通过神经网络训练一遍的次数。每一个 epoch 都包括了一次完整的前向传播和一次完整的后向传播,并且使用了训练集中的所有数据。例如,如果你有 1000 个训练样本,你的 batch size 是 100,那么完成一个 epoch 需要 10 个 iterations(迭代),因为每次迭代处理 100 个样本。多个 epochs 的训练有助于模型学习更多的特征和模式,但是也有过拟合的风险,即模型在训练数据上表现很好,但在未见过的新数据上表现不佳。
# 若构型为孤立原子,可以添加 config_type=IsolatedAtom
1
Lattice="100.0 0.0 0.0 0.0 100.0 0.0 0.0 0.0 100.0" Properties=species:S:1:pos:R:3:forces:R:3 config_type=IsolatedAtom energy=-10.707211383396714 pbc="T T T"
H 0.00000000 0.00000000 0.00000000 -0.00000000 -0.00000000 -0.00000000
MACE-MP0 tuning 命令(王阳帅师兄)
mace_run_train \
--name="MACE" \
--foundation_model="small" \
--train_file="train.xyz" \
--valid_fraction=0.05 \
--test_file="test.xyz" \
--energy_weight=1.0 \
--forces_weight=1.0 \
--E0s="average" \
--lr=0.01 \
--scaling="rms_forces_scaling" \
--batch_size=2 \
--max_num_epochs=6 \
--ema \
--ema_decay=0.99 \
--amsgrad \
--default_dtype="float64" \
--device=cuda \
--seed=3